Beljakovine poganjajo številne biološke procese, kot so encimska aktivnost, molekularni transport in celična podpora. Tridimenzionalna struktura proteina omogoča vpogled v njegovo funkcijo in interakcijo z drugimi biomolekulami. Eksperimentalne metode za določanje strukture beljakovin, kot sta rentgenska kristalografija in NMR spektroskopija, so drage in dolgotrajne.
Nasprotno pa lahko nedavno razvite računalniške metode hitro in natančno napovejo strukturo proteina iz njegovega aminokislinskega zaporedja. Te metode so kritične za beljakovine, ki jih je težko eksperimentalno preučiti, kot so membranski proteini, tarče številnih zdravil. En dobro znan primer tega je AlphaFold, algoritem, ki temelji na globokem učenju, ki je znan po svojih natančnih napovedih.
ESMFold je še ena zelo natančna, na globokem učenju temelječa metoda, razvita za napovedovanje strukture beljakovin iz njihovega aminokislinskega zaporedja. ESMFold uporablja model velikega proteinskega jezika (pLM) kot hrbtenico in deluje od konca do konca. Za razliko od AlphaFold2 ne potrebuje iskanja oz Poravnava več zaporedij (MSA), niti se ne zanaša na zunanje baze podatkov za ustvarjanje napovedi. Namesto tega je razvojna ekipa usposobila model na milijonih proteinskih zaporedij iz UniRefa. Med treningom je model razvil vzorce pozornosti, ki elegantno predstavljajo evolucijske interakcije med aminokislinami v zaporedju. Ta uporaba pLM namesto MSA omogoča do 60-krat hitrejše čase napovedovanja kot drugi najsodobnejši modeli.
V tej objavi uporabljamo vnaprej pripravljen model ESMFold iz Hugging Face with Amazon SageMaker za napovedovanje strukture težke verige trastuzumabA monoklonsko protitelo prvi razvil Genentech za zdravljenje HER2-pozitivni rak dojke. Hitro napovedovanje strukture tega proteina bi lahko bilo koristno, če bi raziskovalci želeli preizkusiti učinek sprememb zaporedja. To bi lahko vodilo do boljšega preživetja bolnikov ali manj stranskih učinkov.
Ta objava ponuja primer zvezka Jupyter in povezanih skriptov v nadaljevanju GitHub repozitorij.
Predpogoji
Priporočamo, da ta primer zaženete v Amazon SageMaker Studio prenosnik izvajanje slike PyTorch 1.13 Python 3.9, optimizirane za CPE, na vrsti primerka ml.r5.xlarge.
Vizualizirajte eksperimentalno strukturo trastuzumaba
Za začetek uporabimo biopython knjižnico in pomožni skript za prenos strukture trastuzumaba iz Banka podatkov o beljakovinah RCSB:
Nato uporabimo py3Dmol knjižnica za vizualizacijo strukture kot interaktivno 3D vizualizacijo:
Naslednja slika predstavlja 3D proteinsko strukturo 1N8Z iz Protein Data Bank (PDB). Na tej sliki je lahka veriga trastuzumaba prikazana v oranžni barvi, težka veriga je modra (s spremenljivo regijo v svetlo modri barvi), antigen HER2 pa je zelen.
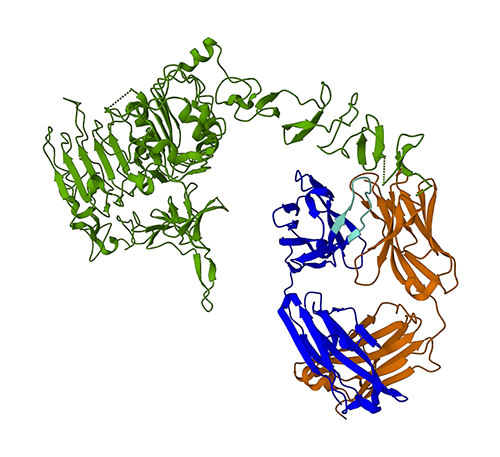
Najprej bomo uporabili ESMFold za napovedovanje strukture težke verige (verige B) iz njenega aminokislinskega zaporedja. Nato bomo primerjali napoved z eksperimentalno določeno strukturo, prikazano zgoraj.
Predvidite strukturo težke verige trastuzumaba iz njegovega zaporedja z uporabo ESMFold
Uporabimo model ESMFold, da napovemo strukturo težke verige in jo primerjamo z eksperimentalnim rezultatom. Za začetek bomo uporabili vnaprej izdelano okolje prenosnika v Studiu, ki ima več pomembnih knjižnic, kot je PyTorch, vnaprej nameščen. Čeprav bi lahko uporabili pospešeno vrsto instance za izboljšanje učinkovitosti naše analize prenosnika, bomo namesto tega uporabili nepospešeno instanco in zagnali predvidevanje ESMFold na CPE.
Najprej naložimo predhodno usposobljen model ESMFold in tokenizer iz Hugging Face Hub:
Nato kopiramo model v našo napravo (v tem primeru CPE) in nastavimo nekaj parametrov modela:
Da bi proteinsko zaporedje pripravili za analizo, ga moramo tokenizirati. To prevede simbole aminokislin (EVQLV…) v numerično obliko, ki jo lahko razume model ESMFold (6,19,5,10,19,…):
Nato kopiramo tokenizirani vnos v način, naredimo napoved in shranimo rezultat v datoteko:
To traja približno 3 minute pri nepospešeni vrsti primerka, kot je r5.
Natančnost napovedi ESMFold lahko preverimo tako, da jo primerjamo z eksperimentalno strukturo. To naredimo z uporabo US-uskladi orodje, ki ga je razvil Zhang Lab na Univerzi v Michiganu:
| PDBchain1 | PDBchain2 | TM-rezultat |
| podatki/napoved.pdb:A | podatki/eksperimentalno.pdb:B | 0.802 |
O rezultat modeliranja predloge (TM-score) je metrika za ocenjevanje podobnosti beljakovinskih struktur. Rezultat 1.0 pomeni popolno ujemanje. Ocene nad 0.7 kažejo, da imajo beljakovine enako strukturo hrbtenice. Ocene nad 0.9 pomenijo, da so beljakovine funkcionalno zamenljivi za nadaljnjo uporabo. V našem primeru doseganja TM-Score 0.802 bi bila napoved ESMFold verjetno primerna za aplikacije, kot je točkovanje strukture ali poskusi vezave liganda, vendar morda ne bi bila primerna za primere uporabe, kot je molekularna zamenjava ki zahtevajo izjemno visoko natančnost.
Ta rezultat lahko potrdimo z vizualizacijo poravnanih struktur. Obe strukturi kažeta visoko, vendar ne popolno stopnjo prekrivanja. Napovedi strukture beljakovin so hitro razvijajoče se področje in številne raziskovalne skupine razvijajo vse natančnejše algoritme!
Namestite ESMFold kot končno točko sklepanja SageMaker
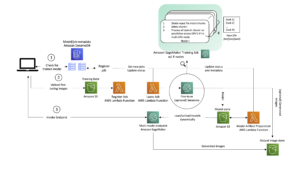
Izvajanje sklepanja o modelu v zvezku je v redu za eksperimentiranje, kaj pa, če morate svoj model integrirati z aplikacijo? Ali cevovod MLOps? V tem primeru je boljša možnost, da svoj model uvedete kot končno točko sklepanja. V naslednjem primeru bomo uvedli ESMFold kot končno točko sklepanja v realnem času SageMaker na pospešeni instanci. Končne točke SageMaker v realnem času zagotavljajo razširljiv, stroškovno učinkovit in varen način za uvajanje in gostovanje modelov strojnega učenja (ML). S samodejnim skaliranjem lahko prilagodite število instanc, ki izvajajo končno točko, da izpolnite zahteve vaše aplikacije, optimizirate stroške in zagotovite visoko razpoložljivost.
Vnaprej zgrajena Posoda SageMaker za Hugging Face omogoča preprosto uvajanje modelov globokega učenja za pogoste naloge. Vendar pa moramo za nove primere uporabe, kot je napoved strukture beljakovin, definirati po meri inference.py skript za nalaganje modela, izvajanje napovedi in oblikovanje izhoda. Ta skript vključuje večino iste kode, ki smo jo uporabili v našem zvezku. Ustvarjamo tudi a requirements.txt datoteko za definiranje nekaterih odvisnosti Pythona za našo končno točko. Datoteke, ki smo jih ustvarili, si lahko ogledate v GitHub repozitorij.
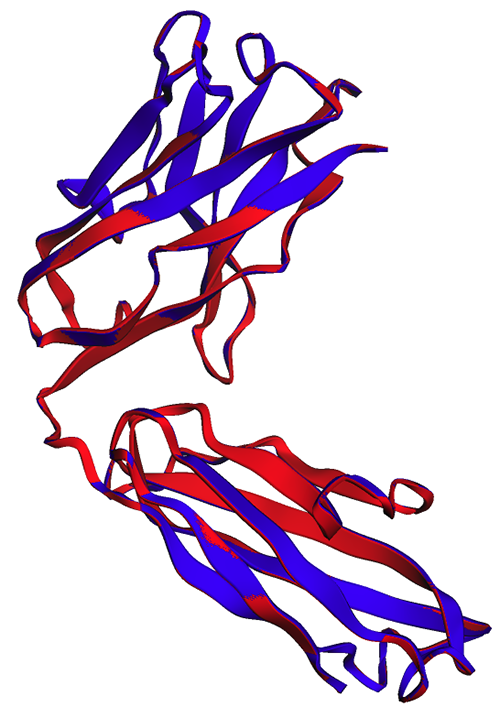
Na naslednji sliki sta eksperimentalna (modra) in predvidena (rdeča) struktura težke verige trastuzumaba zelo podobni, vendar ne enaki.

Ko ustvarimo potrebne datoteke v code imenik, razmestimo naš model z uporabo SageMakerja HuggingFaceModel razred. To uporablja vnaprej zgrajen vsebnik za poenostavitev postopka uvajanja modelov Hugging Face v SageMaker. Upoštevajte, da lahko ustvarjanje končne točke traja 10 minut ali več, odvisno od razpoložljivosti ml.g4dn vrste primerkov v naši regiji.
Ko je uvedba končne točke končana, lahko znova predložimo zaporedje proteinov in prikažemo prvih nekaj vrstic napovedi:
Ker smo našo končno točko razmestili v pospešeno instanco, naj bi predvidevanje trajalo le nekaj sekund. Vsaka vrstica v rezultatu ustreza enemu atomu in vključuje identiteto aminokisline, tri prostorske koordinate in rezultat pLDDT ki predstavlja zanesljivost napovedi na tej lokaciji.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | ZASEBNOST | PLDDT | ATOM_ID |
| ATOM | 1 | N | G.L.U. | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | G.L.U. | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | G.L.U. | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | G.L.U. | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | G.L.U. | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | G.L.U. | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | G.L.U. | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | G.L.U. | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | G.L.U. | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Z uporabo iste metode kot prej vidimo, da sta napovedi beležnice in končne točke enaki.
| PDBchain1 | PDBchain2 | TM-rezultat |
| podatki/endpoint_prediction.pdb:A | podatki/napoved.pdb:A | 1.0 |
Kot je prikazano na naslednji sliki, napovedi ESMFold, ustvarjene v prenosnem računalniku (rdeče) in na končni točki (modro), kažejo popolno poravnavo.
Čiščenje
Da bi se izognili nadaljnjim stroškom, izbrišemo našo končno točko sklepanja in testne podatke:
Povzetek
Računalniška napoved strukture beljakovin je kritično orodje za razumevanje delovanja beljakovin. Poleg osnovnih raziskav imajo algoritmi, kot sta AlphaFold in ESMFold, veliko aplikacij v medicini in biotehnologiji. Strukturni vpogled, pridobljen s temi modeli, nam pomaga bolje razumeti, kako biomolekule medsebojno delujejo. To lahko vodi do boljših diagnostičnih orodij in terapij za bolnike.
V tem prispevku prikazujemo, kako razmestiti proteinski jezikovni model ESMFold iz Hugging Face Hub kot razširljivo končno točko sklepanja z uporabo SageMakerja. Za več informacij o uvajanju modelov Hugging Face na SageMaker glejte Uporabite Hugging Face z Amazon SageMaker. Več primerov znanosti o beljakovinah najdete tudi v Odlična analiza beljakovin na AWS GitHub repo. Pustite komentar, če želite videti še kakšne druge primere!
O avtorjih
 Brian Loyal je višji arhitekt rešitev AI/ML v skupini Global Healthcare and Life Sciences pri Amazon Web Services. Ima več kot 17 let izkušenj na področju biotehnologije in strojnega učenja ter strastno pomaga strankam pri reševanju genomskih in proteomskih izzivov. V prostem času uživa v kuhanju in jedi s prijatelji in družino.
Brian Loyal je višji arhitekt rešitev AI/ML v skupini Global Healthcare and Life Sciences pri Amazon Web Services. Ima več kot 17 let izkušenj na področju biotehnologije in strojnega učenja ter strastno pomaga strankam pri reševanju genomskih in proteomskih izzivov. V prostem času uživa v kuhanju in jedi s prijatelji in družino.
 Shamika Ariyawansa je specialist za rešitve AI/ML v skupini Global Healthcare and Life Sciences pri Amazon Web Services. Strastno sodeluje s strankami, da bi pospešil njihovo sprejemanje AI in ML, tako da jim zagotavlja tehnične smernice in jim pomaga inovirati in graditi varne rešitve v oblaku na AWS. Izven službe obožuje smučanje in brezpotja.
Shamika Ariyawansa je specialist za rešitve AI/ML v skupini Global Healthcare and Life Sciences pri Amazon Web Services. Strastno sodeluje s strankami, da bi pospešil njihovo sprejemanje AI in ML, tako da jim zagotavlja tehnične smernice in jim pomaga inovirati in graditi varne rešitve v oblaku na AWS. Izven službe obožuje smučanje in brezpotja.
 Yanjun Qi je višji vodja uporabnih znanosti v laboratoriju za rešitve strojnega učenja AWS. Uvaja inovacije in uporablja strojno učenje, da strankam AWS pomaga pospešiti njihovo umetno inteligenco in sprejemanje oblaka.
Yanjun Qi je višji vodja uporabnih znanosti v laboratoriju za rešitve strojnega učenja AWS. Uvaja inovacije in uporablja strojno učenje, da strankam AWS pomaga pospešiti njihovo umetno inteligenco in sprejemanje oblaka.
- Distribucija vsebine in PR s pomočjo SEO. Okrepite se še danes.
- PlatoAiStream. Podatkovna inteligenca Web3. Razširjeno znanje. Dostopite tukaj.
- Kovanje prihodnosti z Adryenn Ashley. Dostopite tukaj.
- Kupujte in prodajajte delnice podjetij pred IPO s PREIPO®. Dostopite tukaj.
- vir: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :ima
- : je
- :ne
- $GOR
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- O meni
- nad
- AC
- pospeši
- pospešeno
- natančnost
- natančna
- natančno
- doseganju
- dejavnost
- Poleg tega
- Sprejetje
- AI
- AI / ML
- algoritem
- algoritmi
- poravnano
- poravnava
- Prav tako
- Čeprav
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- Analiza
- in
- Še ena
- kaj
- uporaba
- aplikacije
- uporabna
- primerno
- SE
- AS
- Ocenjevanje
- At
- atom
- pozornosti
- Samodejno
- razpoložljivost
- izogniti
- AWS
- Strojno učenje AWS
- Hrbtenica
- Banka
- Osnovni
- BE
- pred
- začetek
- Boljše
- med
- zavezujoče
- biotehnologije
- Modra
- izgradnjo
- vendar
- by
- CAN
- rak
- cartoon
- primeru
- primeri
- Praznoval
- verige
- izzivi
- Stroški
- preveriti
- razred
- Cloud
- sprejem v oblak
- Koda
- barva
- prihaja
- komentar
- Skupno
- primerjate
- primerjavo
- dokončanje
- zaupanje
- Posoda
- kontrast
- ustreza
- stroškovno učinkovito
- stroški
- bi
- ustvarjajo
- ustvaril
- kritično
- Stranke, ki so
- datum
- baze podatkov
- Datum čas
- globoko
- globoko učenje
- Deepmind
- Stopnja
- zahteve
- Odvisno
- razporedi
- razporejeni
- uvajanja
- uvajanje
- Ugotovite,
- določi
- razvili
- razvoju
- Razvoj
- naprava
- težko
- zaslon
- do
- ne
- Ne
- prenesi
- pogon
- Droge
- med
- vsak
- lahka
- učinek
- Učinki
- omogoča
- konec
- Končna točka
- zagotoviti
- okolje
- Primer
- Primeri
- drago
- izkušnje
- Poskusi
- zunanja
- izredno
- Obraz
- družina
- hitreje
- Nekaj
- manj
- Polje
- Slika
- file
- datoteke
- Najdi
- konec
- prva
- po
- za
- format
- prijatelji
- iz
- funkcija
- nadalje
- ustvarjajo
- ustvarila
- GitHub
- Globalno
- Zelen
- Navodila
- Imajo
- he
- zdravstveno varstvo
- težka
- pomoč
- pomoč
- visoka
- njegov
- gostitelj
- Kako
- Kako
- Vendar
- HTML
- HTTPS
- Hub
- HuggingFace
- enako
- identiteta
- if
- slika
- uvoz
- Pomembno
- izboljšanje
- izboljšalo
- in
- vključuje
- Navedite
- označuje
- Podatki
- inovacije
- vhod
- vpogled
- vpogledi
- primer
- Namesto
- integrirati
- interakcijo
- interakcije
- interaktivno
- interaktivni
- v
- IT
- ITS
- lab
- jezik
- velika
- vodi
- učenje
- pustite
- knjižnice
- Knjižnica
- življenje
- Life Sciences
- light
- kot
- Verjeten
- obremenitev
- kraj aktivnosti
- iskanje
- stroj
- strojno učenje
- Znamka
- IZDELA
- upravitelj
- več
- Stave
- Maj ..
- zdravila
- Srečati
- Metoda
- Metode
- meritev
- Michigan
- milijoni
- Minute
- ML
- MLOps
- način
- Model
- modeli
- spremembe
- molekularno
- več
- veliko
- Ime
- potrebno
- Nimate
- NIH
- prenosnik
- roman
- Številka
- predmeti
- zastarela
- of
- on
- ONE
- samo
- deluje
- optimizacijo
- Možnost
- or
- Oranžna
- OS
- Ostalo
- naši
- izhod
- zunaj
- parametri
- strastno
- Bolnik
- bolniki
- vzorci
- popolna
- performance
- plinovod
- platon
- Platonova podatkovna inteligenca
- PlatoData
- prosim
- Prispevek
- potencialno
- napovedati
- napovedano
- napovedovanje
- napoved
- Napovedi
- Pripravimo
- prej
- Postopek
- Procesi
- Beljakovine
- Beljakovine
- zagotavljajo
- zagotavlja
- zagotavljanje
- Python
- pitorha
- Qi
- hitro
- hitro
- hitro razvijajoče se področje
- v realnem času
- Priporočamo
- Rdeča
- okolica
- povezane
- zanašajo
- predstavljajo
- predstavlja
- predstavlja
- zahteva
- Raziskave
- raziskovalci
- povzroči
- ROW
- Run
- tek
- sagemaker
- Sklep SageMaker
- Enako
- Shrani
- razširljive
- skaliranje
- Znanost
- ZNANOSTI
- rezultat
- točkovanje
- skripte
- sekund
- zavarovanje
- glej
- višji
- Zaporedje
- Storitve
- nastavite
- več
- Delite s prijatelji, znanci, družino in partnerji :-)
- je
- shouldnt
- Prikaži
- pokazale
- strani
- Podoben
- poenostavitev
- sam
- Rešitev
- rešitve
- SOLVE
- nekaj
- prostorsko
- specialist
- Spektroskopija
- hitrost
- Začetek
- state-of-the-art
- Korak
- strukturno
- Struktura
- studio
- študija
- taka
- primerna
- podpora
- preživetje
- Bodite
- meni
- Cilji
- Naloge
- skupina
- Skupine
- tehnični
- Test
- kot
- da
- O
- njihove
- Njih
- POTEM
- Tukaj.
- te
- ta
- 3
- Tridimenzionalni
- čas
- zamudno
- krat
- do
- tokenizirati
- tokenizirano
- orodje
- orodja
- baklo
- usposobljeni
- usposabljanje
- transformatorji
- prevoz
- Zdravljenje
- dva
- tip
- Vrste
- razumeli
- razumevanje
- univerza
- za razliko od
- us
- uporaba
- Rabljeni
- uporabo
- POTRDI
- zelo
- Poglej
- vizualizacija
- W
- hotel
- način..
- we
- web
- spletne storitve
- dobro znana
- Kaj
- Wikipedia
- bo
- z
- WordPress
- delo
- deluje
- bi
- x-ray
- Vi
- Vaša rutina za
- zefirnet