Proteiner driver mange biologiske processer, såsom enzymaktivitet, molekylær transport og cellulær støtte. Den tredimensionelle struktur af et protein giver indsigt i dets funktion og hvordan det interagerer med andre biomolekyler. Eksperimentelle metoder til at bestemme proteinstruktur, såsom røntgenkrystallografi og NMR-spektroskopi, er dyre og tidskrævende.
I modsætning hertil kan nyligt udviklede beregningsmetoder hurtigt og præcist forudsige strukturen af et protein ud fra dets aminosyresekvens. Disse metoder er kritiske for proteiner, der er vanskelige at studere eksperimentelt, såsom membranproteiner, målene for mange lægemidler. Et velkendt eksempel på dette er AlphaFold, en dyb læringsbaseret algoritme, der fejres for sine nøjagtige forudsigelser.
ESMFold er en anden meget nøjagtig, deep learning-baseret metode udviklet til at forudsige proteinstruktur ud fra dets aminosyresekvens. ESMFold bruger en stor proteinsprogmodel (pLM) som rygrad og fungerer fra ende til anden. I modsætning til AlphaFold2 behøver den ikke et opslag eller Multiple Sequence Alignment (MSA) trin, og det er heller ikke afhængigt af eksterne databaser til at generere forudsigelser. I stedet trænede udviklingsteamet modellen på millioner af proteinsekvenser fra UniRef. Under træningen udviklede modellen opmærksomhedsmønstre, der elegant repræsenterer de evolutionære interaktioner mellem aminosyrer i sekvensen. Denne brug af en pLM i stedet for en MSA muliggør op til 60 gange hurtigere forudsigelsestider end andre state-of-the-art modeller.
I dette indlæg bruger vi den fortrænede ESMFold-model fra Hugging Face med Amazon SageMaker at forudsige den tunge kædestruktur af trastuzumab, en monoklonalt antistof først udviklet af Genentech til behandling af HER2-positiv brystkræft. Hurtigt at forudsige strukturen af dette protein kunne være nyttigt, hvis forskere ønskede at teste effekten af sekvensændringer. Dette kan potentielt føre til forbedret patientoverlevelse eller færre bivirkninger.
Dette indlæg giver et eksempel på en Jupyter-notesbog og relaterede scripts i det følgende GitHub repository.
Forudsætninger
Vi anbefaler at køre dette eksempel i en Amazon SageMaker Studio notesbog kører det PyTorch 1.13 Python 3.9 CPU-optimerede billede på en ml.r5.xlarge instanstype.
Visualiser den eksperimentelle struktur af trastuzumab
Til at begynde med bruger vi biopython bibliotek og et hjælpescript til at downloade trastuzumab-strukturen fra RCSB Protein Databank:
Dernæst bruger vi py3Dmol bibliotek for at visualisere strukturen som en interaktiv 3D-visualisering:
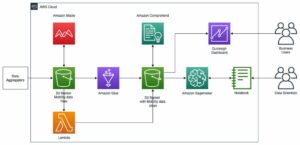
Følgende figur repræsenterer 3D-proteinstrukturen 1N8Z fra Protein Data Bank (PDB). På dette billede vises den lette trastuzumab-kæde i orange, den tunge kæde er blå (med den variable region i lyseblå), og HER2-antigenet er grønt.
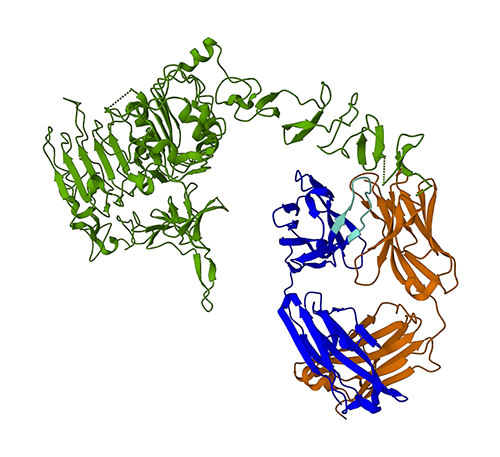
Vi vil først bruge ESMFold til at forudsige strukturen af den tunge kæde (Kæde B) ud fra dens aminosyresekvens. Derefter vil vi sammenligne forudsigelsen med den eksperimentelt bestemte struktur vist ovenfor.
Forudsige trastuzumabs tunge kædestruktur ud fra dens sekvens ved hjælp af ESMFold
Lad os bruge ESMFold-modellen til at forudsige strukturen af den tunge kæde og sammenligne den med det eksperimentelle resultat. Til at starte med bruger vi et forudbygget notebook-miljø i Studio, der kommer med flere vigtige biblioteker, som f.eks. PyTorch, forudinstalleret. Selvom vi kunne bruge en accelereret instanstype til at forbedre ydeevnen af vores notebook-analyse, bruger vi i stedet en ikke-accelereret instans og kører ESMFold-forudsigelsen på en CPU.
Først indlæser vi den fortrænede ESMFold-model og tokenizer fra Krammer Face Hub:
Derefter kopierer vi modellen til vores enhed (CPU i dette tilfælde) og indstiller nogle modelparametre:
For at forberede proteinsekvensen til analyse skal vi tokenisere den. Dette oversætter aminosyresymbolerne (EVQLV...) til et numerisk format, som ESMFold-modellen kan forstå (6,19,5,10,19,...):
Derefter kopierer vi det tokeniserede input til tilstanden, laver en forudsigelse og gemmer resultatet i en fil:
Dette tager omkring 3 minutter på en ikke-accelereret instanstype, som en r5.
Vi kan kontrollere nøjagtigheden af ESMFold-forudsigelsen ved at sammenligne den med den eksperimentelle struktur. Det gør vi ved hjælp af US-Align værktøj udviklet af Zhang Lab ved University of Michigan:
| PDB-kæde1 | PDB-kæde2 | TM-Score |
| data/forudsigelse.pdb:A | data/eksperimentel.pdb:B | 0.802 |
skabelon modellering score (TM-score) er en metrik til vurdering af ligheden mellem proteinstrukturer. En score på 1.0 indikerer et perfekt match. Score over 0.7 indikerer, at proteiner deler den samme rygradsstruktur. Score over 0.9 indikerer, at proteinerne er funktionelt udskiftelige til downstream-brug. I vores tilfælde med at opnå TM-Score 0.802, ville ESMFold-forudsigelsen sandsynligvis være passende til applikationer som strukturscoring eller ligandbindingseksperimenter, men er muligvis ikke egnet til brugssager som f.eks. molekylær udskiftning som kræver ekstrem høj nøjagtighed.
Vi kan validere dette resultat ved at visualisere de tilpassede strukturer. De to strukturer viser en høj, men ikke perfekt, grad af overlap. Forudsigelser af proteinstruktur er et felt i hastig udvikling, og mange forskerhold udvikler stadig mere nøjagtige algoritmer!
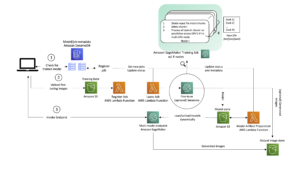
Implementer ESMFold som et SageMaker-slutpunkt
At køre modelinferens i en notesbog er fint til eksperimentering, men hvad nu hvis du har brug for at integrere din model med en applikation? Eller en MLOps pipeline? I dette tilfælde er en bedre mulighed at implementere din model som et slutningsendepunkt. I det følgende eksempel vil vi implementere ESMFold som et SageMaker-slutpunkt for realtidsslutning på en accelereret instans. SageMaker-endepunkter i realtid giver en skalerbar, omkostningseffektiv og sikker måde at implementere og hoste maskinlæringsmodeller (ML). Med automatisk skalering kan du justere antallet af forekomster, der kører endepunktet, for at imødekomme kravene fra din applikation, optimere omkostningerne og sikre høj tilgængelighed.
Den forudbyggede SageMaker beholder til Hugging Face gør det nemt at implementere deep learning-modeller til almindelige opgaver. For nye anvendelsestilfælde som forudsigelse af proteinstruktur er vi dog nødt til at definere en skik inference.py script for at indlæse modellen, køre forudsigelsen og formatere outputtet. Dette script indeholder meget af den samme kode, som vi brugte i vores notesbog. Vi laver også en requirements.txt fil for at definere nogle Python-afhængigheder, som vores slutpunkt skal bruge. Du kan se de filer, vi oprettede i GitHub repository.
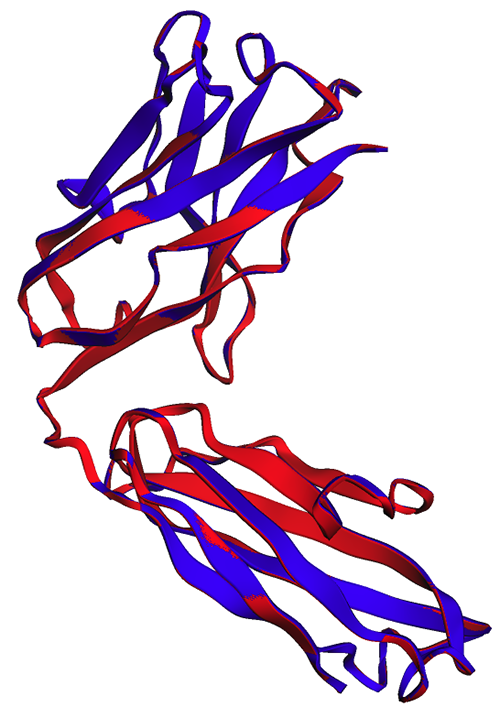
I den følgende figur er de eksperimentelle (blå) og forudsagte (røde) strukturer af den tunge trastuzumab-kæde meget ens, men ikke identiske.

Efter at vi har oprettet de nødvendige filer i code bibliotek, implementerer vi vores model ved hjælp af SageMaker HuggingFaceModel klasse. Dette bruger en forudbygget container til at forenkle processen med at implementere Hugging Face-modeller til SageMaker. Bemærk, at det kan tage 10 minutter eller mere at oprette slutpunktet, afhængigt af tilgængeligheden af ml.g4dn instanstyper i vores region.
Når slutpunktsimplementeringen er færdig, kan vi genindsende proteinsekvensen og vise de første par rækker af forudsigelsen:
Fordi vi implementerede vores endepunkt til en accelereret instans, skulle forudsigelsen kun tage et par sekunder. Hver række i resultatet svarer til et enkelt atom og inkluderer aminosyreidentiteten, tre rumlige koordinater og en pLDDT-score repræsenterer forudsigelsestilliden på det pågældende sted.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | BELÆGNING | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Ved at bruge samme metode som før ser vi, at notebook- og slutpunktsforudsigelserne er identiske.
| PDB-kæde1 | PDB-kæde2 | TM-Score |
| data/endpoint_prediction.pdb:A | data/forudsigelse.pdb:A | 1.0 |
Som observeret i den følgende figur viser ESMFold-forudsigelserne genereret i notebook (rød) og ved slutpunktet (blå) perfekt justering.
Ryd op
For at undgå yderligere gebyrer sletter vi vores slutpunkt- og testdata:
Resumé
Computational protein struktur forudsigelse er et kritisk værktøj til at forstå funktionen af proteiner. Ud over grundforskning har algoritmer som AlphaFold og ESMFold mange anvendelsesmuligheder inden for medicin og bioteknologi. Den strukturelle indsigt, der genereres af disse modeller, hjælper os med bedre at forstå, hvordan biomolekyler interagerer. Dette kan så føre til bedre diagnostiske værktøjer og terapier for patienter.
I dette indlæg viser vi, hvordan man implementerer ESMFold-proteinsprogmodellen fra Hugging Face Hub som et skalerbart inferensendepunkt ved hjælp af SageMaker. For mere information om implementering af Hugging Face-modeller på SageMaker, se Brug Hugging Face med Amazon SageMaker. Du kan også finde flere proteinvidenskabelige eksempler i Fantastisk proteinanalyse på AWS GitHub repo. Efterlad os en kommentar, hvis der er andre eksempler, du gerne vil se!
Om forfatterne
 Brian Loyal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mere end 17 års erfaring inden for bioteknologi og maskinlæring og brænder for at hjælpe kunder med at løse genomiske og proteomiske udfordringer. I sin fritid nyder han at lave mad og spise sammen med sine venner og familie.
Brian Loyal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mere end 17 års erfaring inden for bioteknologi og maskinlæring og brænder for at hjælpe kunder med at løse genomiske og proteomiske udfordringer. I sin fritid nyder han at lave mad og spise sammen med sine venner og familie.
 Shamika Ariyawansa er AI/ML Specialist Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han arbejder lidenskabeligt med kunder for at accelerere deres AI- og ML-adoption ved at give teknisk vejledning og hjælpe dem med at innovere og bygge sikre cloud-løsninger på AWS. Uden for arbejdet elsker han at stå på ski og offroad.
Shamika Ariyawansa er AI/ML Specialist Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han arbejder lidenskabeligt med kunder for at accelerere deres AI- og ML-adoption ved at give teknisk vejledning og hjælpe dem med at innovere og bygge sikre cloud-løsninger på AWS. Uden for arbejdet elsker han at stå på ski og offroad.
 Yanjun Qi er Senior Applied Science Manager hos AWS Machine Learning Solution Lab. Hun innoverer og anvender maskinlæring for at hjælpe AWS-kunder med at fremskynde deres AI og cloud-adoption.
Yanjun Qi er Senior Applied Science Manager hos AWS Machine Learning Solution Lab. Hun innoverer og anvender maskinlæring for at hjælpe AWS-kunder med at fremskynde deres AI og cloud-adoption.
- SEO Powered Content & PR Distribution. Bliv forstærket i dag.
- PlatoAiStream. Web3 Data Intelligence. Viden forstærket. Adgang her.
- Udmøntning af fremtiden med Adryenn Ashley. Adgang her.
- Køb og sælg aktier i PRE-IPO-virksomheder med PREIPO®. Adgang her.
- Kilde: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :har
- :er
- :ikke
- $OP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Om
- over
- AC
- fremskynde
- accelereret
- nøjagtighed
- præcis
- præcist
- opnå
- aktivitet
- Desuden
- Vedtagelse
- AI
- AI / ML
- algoritme
- algoritmer
- justeret
- tilpasning
- også
- Skønt
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- analyse
- ,
- En anden
- enhver
- Anvendelse
- applikationer
- anvendt
- passende
- ER
- AS
- Vurdering
- At
- atom
- opmærksomhed
- Automatisk Ur
- tilgængelighed
- undgå
- AWS
- AWS maskinindlæring
- Backbone
- Bank
- grundlæggende
- BE
- før
- begynde
- Bedre
- mellem
- binding
- bioteknologi
- Blå
- bygge
- men
- by
- CAN
- Kræft
- tegneserie
- tilfælde
- tilfælde
- fejret
- kæde
- udfordringer
- afgifter
- kontrollere
- klasse
- Cloud
- cloud adoption
- kode
- farve
- kommer
- KOMMENTAR
- Fælles
- sammenligne
- sammenligne
- fuldføre
- tillid
- Container
- kontrast
- svarer
- omkostningseffektiv
- Omkostninger
- kunne
- skabe
- oprettet
- kritisk
- Kunder
- data
- databaser
- dato tid
- dyb
- dyb læring
- DeepMind
- Degree
- krav
- Afhængigt
- indsætte
- indsat
- implementering
- implementering
- Bestem
- bestemmes
- udviklet
- udvikling
- Udvikling
- enhed
- svært
- Skærm
- do
- gør
- Er ikke
- downloade
- køre
- Narkotika
- i løbet af
- hver
- let
- effekt
- effekter
- muliggør
- ende
- Endpoint
- sikring
- Miljø
- eksempel
- eksempler
- dyrt
- erfaring
- eksperimenter
- ekstern
- ekstremt
- Ansigtet
- familie
- hurtigere
- få
- færre
- felt
- Figur
- File (Felt)
- Filer
- Finde
- ende
- Fornavn
- efter
- Til
- format
- venner
- fra
- funktion
- yderligere
- generere
- genereret
- GitHub
- Global
- Grøn
- vejledning
- Have
- he
- sundhedspleje
- tunge
- hjælpe
- hjælpe
- Høj
- hans
- host
- Hvordan
- How To
- Men
- HTML
- HTTPS
- Hub
- KrammerFace
- identisk
- Identity
- if
- billede
- importere
- vigtigt
- Forbedre
- forbedret
- in
- omfatter
- angiver
- angiver
- oplysninger
- innovere
- indgang
- indsigt
- indsigt
- instans
- i stedet
- integrere
- interagere
- interaktioner
- interaktiv
- interagerer
- ind
- IT
- ITS
- lab
- Sprog
- stor
- føre
- læring
- Forlade
- biblioteker
- Bibliotek
- Livet
- Life Sciences
- lys
- ligesom
- Sandsynlig
- belastning
- placering
- kig op
- maskine
- machine learning
- lave
- maerker
- leder
- mange
- Match
- Kan..
- medicin
- Mød
- metode
- metoder
- metrisk
- Michigan
- millioner
- minutter
- ML
- MLOps
- tilstand
- model
- modeller
- Modifikationer
- molekylær
- mere
- meget
- navn
- nødvendig
- Behov
- NIH
- notesbog
- roman
- nummer
- objekter
- forældede
- of
- on
- ONE
- kun
- opererer
- optimering
- Option
- or
- Orange
- OS
- Andet
- vores
- output
- uden for
- parametre
- lidenskabelige
- patient
- patienter
- mønstre
- perfekt
- ydeevne
- pipeline
- plato
- Platon Data Intelligence
- PlatoData
- Vær venlig
- Indlæg
- potentielt
- forudsige
- forudsagde
- forudsige
- forudsigelse
- Forudsigelser
- Forbered
- tidligere
- behandle
- Processer
- Protein
- Proteiner
- give
- giver
- leverer
- Python
- pytorch
- Qi
- hurtigt
- hurtigt
- felt i hastig udvikling
- realtid
- anbefaler
- Rød
- region
- relaterede
- stole
- repræsentere
- repræsenterer
- repræsenterer
- kræver
- forskning
- forskere
- resultere
- RÆKKE
- Kør
- kører
- sagemaker
- SageMaker Inference
- samme
- Gem
- skalerbar
- skalering
- Videnskab
- VIDENSKABER
- score
- scoring
- scripts
- sekunder
- sikker
- se
- senior
- Sequence
- Tjenester
- sæt
- flere
- Del
- hun
- bør
- Vis
- vist
- side
- lignende
- forenkle
- enkelt
- løsninger
- Løsninger
- SOLVE
- nogle
- rumlige
- specialist
- Spektroskopi
- hastighed
- starte
- state-of-the-art
- Trin
- strukturel
- struktur
- Studio
- Studere
- sådan
- egnede
- support
- overlevelse
- Tag
- tager
- mål
- opgaver
- hold
- hold
- Teknisk
- prøve
- end
- at
- deres
- Them
- derefter
- Der.
- Disse
- denne
- tre
- tredimensionale
- tid
- tidskrævende
- gange
- til
- tokenisere
- tokeniseret
- værktøj
- værktøjer
- fakkel
- uddannet
- Kurser
- transformers
- transportere
- behandling
- to
- typen
- typer
- forstå
- forståelse
- universitet
- I modsætning til
- us
- brug
- anvendte
- ved brug af
- VALIDATE
- meget
- Specifikation
- visualisering
- W
- ønskede
- Vej..
- we
- web
- webservices
- Kendt
- Hvad
- Wikipedia
- vilje
- med
- WordPress
- Arbejde
- virker
- ville
- røntgen
- Du
- Din
- zephyrnet