Introduksjon
Oppdagelsen under Human Genome Project på begynnelsen av 2000-tallet at vi mennesker bare har rundt 20,000 XNUMX proteinkodende gener - omtrent like mange som den lille jordlevende nematodeormen, og mindre enn halvparten så mange som risplanten - kom som et sjokk . Dette slaget mot vår stolthet ble imidlertid mildnet av ideen om at det menneskelige genomet er rikt på regulatoriske forbindelser. Genene våre samhandler i et tett nettverk, der biter av DNA og molekylene de koder for (RNA og proteiner) kontrollerer «uttrykket» av andre gener, og påvirker om de lager deres respektive RNA og proteiner. For å forstå det menneskelige genomet, trengte vi å forstå denne prosessen med genregulering.
Den oppgaven viser seg imidlertid å være mye vanskeligere enn å dekode sekvensen til genomet.
Opprinnelig ble det mistenkt at genregulering var et enkelt spørsmål om at ett genprodukt fungerte som en av/på-bryter for et annet gen, på digital måte. På 1960-tallet belyste de franske biologene François Jacob og Jacques Monod først en genregulerende prosess i mekanistisk detalj: In Escherichia coli bakterier, når et repressorprotein binder seg til et bestemt segment av DNA, blokkerer det transkripsjonen og translasjonen av en tilstøtende serie gener som koder for enzymer for å fordøye sukkerlaktosen. Denne reguleringskretsen, som Monod og Jacob kalte innsjø operon, har en pen, gjennomsiktig logikk.
Men genregulering i komplekse metazoer - dyr som mennesker, med komplekse eukaryote celler - ser vanligvis ikke ut til å fungere på denne måten. I stedet involverer det en gjeng med molekyler, inkludert proteiner, RNA og biter av DNA fra hele et kromosom, som på en eller annen måte samarbeider for å kontrollere uttrykket av et gen.
Det er ikke bare at denne reguleringsprosessen i eukaryoter har flere spillere enn det som vanligvis sees i bakterier og andre enkle, prokaryote celler; det ser ut til å være en kategorisk annerledes prosess, og en mer uklar prosess.
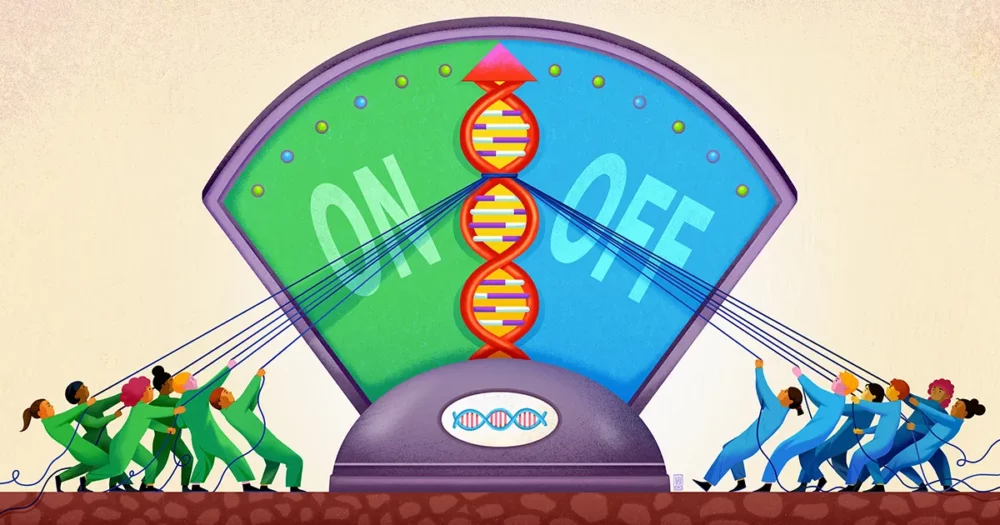
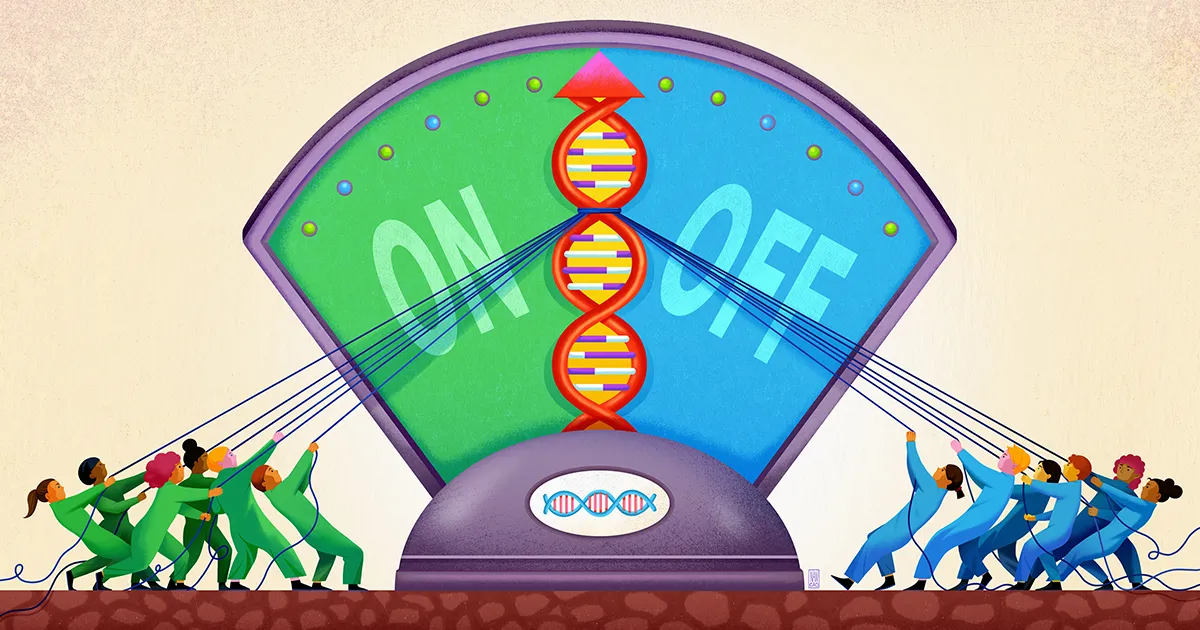
Et team ved Stanford University, ledet av biofysikeren og bioingeniøren Polly Fordyce, ser nå ut til å ha avdekket en del av denne uklare modusen for genregulering. Deres arbeid, publisert i september i fjor Vitenskap, antyder at DNA i nærheten av et gen fungerer som en slags grunn brønn for å fange forskjellige regulatoriske molekyler, og holde dem klare for handling, slik at de ved behov kan legge sin stemme til beslutningen om hvorvidt de skal aktivere genet.
Introduksjon
Disse reguleringsbrønnene er laget av desidert merkelige strekninger med DNA. De består av sekvenser der et kort DNA-stykke, fra ett til seks basepar langt, gjentas mange ganger. Titalls kopier av disse "korte tandem-repetisjonene" (STR-ene) kan settes sammen i disse sekvensene, som det samme lille "ordet" skrevet igjen og igjen.
STR-er er rikelig i det menneskelige genomet: De utgjør omtrent 5 % av alt vårt DNA. De ble en gang antatt å være klassiske eksempler på "søppel"-DNA fordi en repeterende DNA-"tekst" som bare består av STR-er, ikke kan inneholde på langt nær så mye meningsfull informasjon som for eksempel den uregelmessige rekkefølgen av bokstaver som utgjør en setning i denne artikkel.
Og likevel er STR-er tydeligvis ikke ubetydelige: De har vært knyttet til plager som Huntingtons sykdom, spinobulbar muskelatrofi, Crohns sykdom og noen kreftformer. I løpet av de siste par tiårene har det samlet seg bevis for at de på en eller annen måte kan forbedre eller hemme genregulering. Mysteriet var hvordan de kunne være så kraftige med så lite informasjonsinnhold.
Komplekse kontroller for komplekse celler
For å forstå hvordan STR-er passer inn i det store bildet av genregulering, la oss ta et skritt tilbake. Gener er rutinemessig flankert av biter av DNA som ikke koder for RNA eller protein, men som har regulatoriske funksjoner. Bakterielle gener har "promotor"-regioner der polymeraseenzymer kan binde seg for å starte transkripsjonen av tilstøtende DNA til RNA. De har også rutinemessig "operatør"-regioner, der repressorproteiner kan binde seg for å blokkere transkripsjon, og slå av et gen, som i innsjø operon.
Hos mennesker og andre eukaryoter kan de regulatoriske sekvensene være flere, forskjellige - og forvirrende. Regioner kalt forsterkere, for eksempel, påvirker sannsynligheten for at et gen vil bli transkribert. Forsterkere er ofte målet for proteiner kalt transkripsjonsfaktorer, som kan binde seg for å øke eller hemme genuttrykk. Merkelig nok er noen forsterkere titusenvis av basepar borte fra genene de regulerer, og bringes bare nær dem gjennom den fysiske omorganiseringen av DNA-løkkene i et pakket kromosom.
Eukaryotisk genregulering involverer vanligvis disse mange forskjellige regulatoriske blokkene av DNA, sammen med en eller flere transkripsjonsfaktorer og andre molekyler, som alle samles rundt et gen som en komité som er satt sammen for å bestemme hva den skal gjøre. De samles i en løs, tett klynge.
Ofte ser det heller ikke ut til at de molekylære deltakerne samhandler gjennom de svært selektive "lås og nøkkel"-parringene som er vanlige i molekylærbiologi. De er i stedet mye mindre kresne, samhandler ganske svakt og uselektivt, som om de vandrer rundt og innleder korte samtaler med hverandre.
Faktisk har det vært noe av et mysterium hvordan transkripsjonsfaktorer binder seg til DNA i eukaryoter. Det ble lenge antatt at en del av en transkripsjonsfaktor må samsvare nøye med en bindende "motiv"-sekvens i DNA, som bitene i et puslespill. Men selv om noen slike motiver har blitt identifisert, samsvarer ikke deres tilstedeværelse alltid veldig godt med hvor forskere finner transkripsjonsfaktorer som fester seg til DNA i cellene. Noen ganger henger transkripsjonsfaktorer igjen i områder uten motiver, mens noen motiver som virker som om de skulle binde transkripsjonsfaktorer sterkt forblir tomme.
"Tradisjonelt i genomikk har målet vært å klassifisere genomiske steder på en [binær] måte som enten 'bundet' eller 'ubundet'" av transkripsjonsfaktorer, sa Fordyce. "Men bildet er mye mer nyansert enn som så." De enkelte medlemmene av disse genregulerende "komiteene" ser ikke ut til å alltid være tilstede for eller fraværende fra møtene deres, men snarere har forskjellige sannsynligheter for å være der eller ikke.
Tendensen til genregulering i eukaryoter til å stole på så mange forskjellige svake interaksjoner mellom store molekylære komplekser "er en av tingene som gjør det notorisk vanskelig å få tak på teoretisk," sa biofysikeren Thomas Kuhlman fra University of California, Riverside, som skrev en kommentar på Fordyce-laboratoriets papir for Vitenskap. Det er et dypt puslespill hvordan, ut av denne tilsynelatende kaotiske prosessen, dukker det opp presise beslutninger om å slå gener på og av.
Utover den mystiske uklare logikken i den beslutningsprosessen, er det også spørsmålet om hvordan alle komitémedlemmene finner veien til det rette rommet - og deretter blir der. Molekyler beveger seg vanligvis rundt i cellen ved diffusjon, påvirket av alle de andre omkringliggende molekylene, som vann, og vandrer i tilfeldige retninger. Vi kan forvente at disse løse komiteene går fra hverandre for raskt til å gjøre reguleringsjobben sin.
Det, tror Fordyce og hennes kolleger, er der STR-ene kommer inn. STR-er er slående vanlige innenfor enhancer-steder på DNA. I papiret deres argumenterer forskerne for at STR-ene fungerer som klebrige flekker som kaller opp transkripsjonsfaktorer og stopper dem fra å forville seg.
Finjustering av klebrigheten
Fordyces gruppe undersøkte systematisk hvordan forskjeller i STR-sekvens påvirker bindingen av transkripsjonsfaktorer til et bindende motiv. De så på to faktorer - en fra gjær, en fra mennesker - som holder seg til et bestemt seks-base-motiv. Forskerne målte både styrken (eller affiniteten) til den bindingen og hastigheten som transkripsjonsfaktorene blir sittende fast og løs (kinetikk) når motivet er flankert av en STR i stedet for en tilfeldig sekvens. Til sammenligning så de på hvor lett faktorene binder seg til STR alene og til en helt tilfeldig DNA-sekvens.
"En av de største utfordringene i dette feltet er å skille ut de utallige variablene som påvirker [transkripsjonsfaktor] binding i en bestemt posisjon av genomet," sa David Suter, en molekylærbiolog ved Swiss Federal Institute of Technology Lausanne i Sveits. DNA-form, nærhet til andre DNA-segmenter og fysisk spenning i DNA-molekylene kan alle spille en rolle i transkripsjonsfaktorbinding. Verdiene til disse parameterne varierer sannsynligvis ved hver posisjon i genomet, og kanskje også mellom celletyper og innenfor en enkelt celle over tid på en gitt posisjon. "Dette er et stort rom med ukjente variabler som er svært vanskelig å kvantifisere," sa Suter.
Introduksjon
Det er derfor godt kontrollerte eksperimenter som de fra Stanford-teamet er så nyttige, la Kuhlman til. Vanligvis, når forskere trenger å måle svake interaksjoner som disse, har de to valg: De kan gjøre noen få svært detaljerte, ekstremt presise målinger og generalisere fra dem, eller de kan ta svært mange raske og skitne målinger og bruke matematisk komplekse. statistiske metoder for å utlede resultater. Men Fordyce og hennes kolleger, sa Kuhlman, brukte en automatisert, mikrofluidisk brikkebasert prosedyre for å ta presise målinger under eksperimenter med høy gjennomstrømning "for å få det beste fra begge verdener."
Stanford-teamet fant at forskjellige STR-sekvenser kan endre bindingsaffinitetene til transkripsjonsfaktorer til DNA med så mye som en faktor på 70; de har noen ganger større innvirkning på transkripsjonsfaktorbinding enn å endre sekvensen til selve bindingsmotivet. Og effektene var forskjellige for de to forskjellige transkripsjonsfaktorene de så på.
Så STR-er ser ut til å finjustere evnen til transkripsjonsfaktorer til å forankre på et DNA-sted og dermed regulere et gen. Men hvordan, nøyaktig?
Et venterom nær et gen
Forskerne skjønte at den delen av en transkripsjonsfaktor som binder DNA kan samhandle svakt med en STR, med den nøyaktige styrken til den affiniteten avhengig av STR-sekvensen. Fordi slik binding er svak, vil den ikke ha mye spesifisitet. Men hvis en transkripsjonsfaktor blir løst grepet og frigjort av en STR igjen og igjen, er den kumulative effekten å holde transkripsjonsfaktoren i nærheten av genet slik at det er mer sannsynlig at den binder seg sikkert til motivregionen om nødvendig.
Fordyce og hennes kolleger spådde at STR-er dermed fungerer som en "lobby" eller brønn hvor transkripsjonsfaktorer kan samle seg, men forbigående, nær et regulatorisk bindingssted. "Den repeterende naturen til en STR forsterker den svake effekten av et enkelt bindingssted den er laget av," sa Connor Horton, den første forfatteren på studien, som nå er doktorgradsstudent ved University of California, Berkeley.
Omvendt, la han til, kan noen STR-er også virke for å trekke transkripsjonsfaktorer bort fra regulatoriske sekvenser, og suge opp transkripsjonsfaktorer andre steder som en svamp. På denne måten kan de hemme genuttrykk.
Arbeidet, sa Suter, "viser overbevisende at STR-er direkte påvirker bindingen av transkripsjonsfaktorer in vitro." Dessuten brukte Stanford-teamet en maskinlæringsalgoritme for å vise at effektene som ble sett i in vitro-eksperimentene deres også ser ut til å forekomme i levende celler (det vil si in vivo).
Men Robert Tjian, en biokjemiker ved Berkeley og en etterforsker ved Howard Hughes Medical Institute, mener det kan være for tidlig å være sikker på hvilken innflytelse en gitt STR-transkripsjonsfaktorkombinasjon har på genuttrykk i virkelige celler.
Tjian, Xavier Darzacq og kollegene deres i laboratoriet de driver sammen på Berkeley er enige om at STR-er ser ut til å tilby en måte å konsentrere transkripsjonsfaktorer i nærheten av genregulerende steder. Men uten å vite hvor nærme faktorene må være for å aktivere transkripsjon, er det vanskelig å forstå den funksjonelle betydningen av dette resultatet. Tjian sa at han gjerne vil se om introduksjon av en STR i en levende celle forutsigbart påvirker uttrykket av et målgen. For øyeblikket, sa han, er han "ikke overbevist om at STR-er nødvendigvis kommer til å være et viktig aspekt av [regulatoriske] mekanismer in vivo."
En kombinatorisk grammatikk
Et dvelende puslespill er hvordan en slik mekanisme pålitelig gir den typen presis genregulering som celler trenger, siden både styrken og selektiviteten til transkripsjonsfaktorbinding i STR-brønnene er svak. Fordyce tror at en slik spesifisitet av påvirkning kan komme fra mange kilder - ikke bare fra forskjeller i STR-sekvensene, men også fra samarbeidende interaksjoner mellom transkripsjonsfaktorer og andre proteiner involvert i regulering.
Gitt alt dette, sa Horton, er det ikke klart at det vil være enkelt å forutsi effekten av en gitt STR-transkripsjonsfaktorkombinasjon på ekspresjonen av et gen. Logikken i prosessen er uklar. Og "grammatikken" av påvirkningen er sannsynligvis kombinatorisk, la Horton til: Utfallet avhenger av forskjellige kombinasjoner av transkripsjonsfaktorer og andre molekyler.
Stanford-teamet tror at kanskje 90 % av transkripsjonsfaktorene er følsomme for STR-er, men at det er mange flere typer transkripsjonsfaktorer i det menneskelige genomet enn det er typer STR-er. "Å mutere en STR-sekvens kan påvirke bindingen av 20 forskjellige transkripsjonsfaktorer i den celletypen, noe som fører til en generell reduksjon i transkripsjon av det nærliggende genet uten å implisere noen spesifikk transkripsjonsfaktor," sa Horton.
Så faktisk er Stanford-teamet enig med Tjian i at genregulering i levende celler ikke kommer til å bli drevet av en enkelt, enkel mekanisme. Snarere kan transkripsjonsfaktorer, deres DNA-bindingsseter og andre regulatoriske molekyler samles til tette samlinger som utøver sin innflytelse kollektivt.
"Det er nå flere eksempler som støtter ideen om at DNA-elementer kan samle transkripsjonsfaktorer til det punktet hvor de danner kondensat med kofaktorer," sa Richard Young, en cellebiolog ved Whitehead Institute ved Massachusetts Institute of Technology. Enhancers binder mange transkripsjonsfaktorer for å produsere den trengselen. STR-er kan være en ingrediens som hjelper med å samle transkripsjonsfaktorer til å samle seg nær et gen, men de vil ikke være hele historien.
Hvorfor regulere gener på denne kompliserte måten, i stedet for å stole på den typen sterke og spesifikke interaksjoner mellom regulatoriske proteiner og DNA-steder som dominerer i prokaryoter? Det er mulig at slik uklarhet er det som i det hele tatt gjorde store komplekse metazoer mulig.
For å være levedyktige arter må organismer være i stand til å utvikle seg og tilpasse seg endrede omstendigheter. Hvis cellene våre var avhengige av et enormt, men strengt foreskrevet nettverk av genregulerende interaksjoner, ville det være vanskelig å gjøre noen endringer i det uten å forstyrre hele innretningen, akkurat som en sveitsisk klokke vil gripe hvis vi fjerner (eller til og med litt fortrenger) noen av dens utallige tannhjul. Hvis de regulatoriske molekylære interaksjonene er løse og ganske uspesifikke, er det imidlertid nyttig slakk i systemet – akkurat som en komité generelt kan komme til en god avgjørelse selv om et av medlemmene er sykemeldt.
Fordyce bemerker at i prokaryoter som bakterier kan det være relativt enkelt for transkripsjonsfaktorer å finne bindingsstedene deres fordi genomet som skal søkes i er mindre. Men det blir vanskeligere ettersom genomet blir større. I de store genomene til eukaryoter, "kan du ikke lenger tolerere risikoen for at du forbigående blir sittende fast på et 'feil' bindingssted," sa Fordyce, fordi det ville kompromittere evnen til å reagere raskt på skiftende miljøforhold.
Dessuten er STR i seg selv svært utviklingsdyktige. En forlengelse eller forkorting av sekvensen deres, eller en endring av størrelsen og dybden til "transkripsjonsfaktorbrønnen", kan lett oppstå gjennom uhell i DNA-replikasjon eller reparasjon, eller gjennom seksuell rekombinasjon av kromosomene. For Fordyce antyder det at STR-er "derfor kan tjene som råstoffet for å utvikle nye reguleringselementer og finjustere eksisterende reguleringsmoduler for sensitive transkripsjonsprogrammer," for eksempel de som styrer utviklingen av dyr og planter.
Kraften til svake interaksjoner
Slike hensyn får molekylærbiologer til å gi mye mer oppmerksomhet til svake og relativt uselektive interaksjoner i genomet. Mange av disse involverer proteiner som, i stedet for å ha en fast og presis struktur, er løse og floppy - "iboende uorden," som biokjemikere sa det. Hvis proteiner bare virket gjennom stive strukturelle domener, forklarte Young, ville det begrense ikke bare hvor godt regulatoriske systemer kunne utvikle seg, men også hvilke typer dynamisk regulering man ser i livet. "Du vil ikke finne en levende organisme - eller til og med et virus - som fungerer med bare stabile strukturelle elementer som de i en sveitsisk klokke," sa Young.
Kanskje snublet evolusjonen bare over STR-er som en del av en så kompleks, men til slutt mer effektiv løsning på genregulering i eukaryoter. STR-er i seg selv kan oppstå på flere måter - for eksempel gjennom feil i DNA-replikasjon eller aktiviteten til DNA-segmenter kalt transponerbare elementer som lager kopier av seg selv gjennom genomet.
"Det skjedde at de resulterende svake interaksjonene mellom proteiner og de repeterende sekvensene var noe som kunne ... gi selektiv fordel til cellene der det skjedde," sa Kuhlman. Hans gjetning er at denne uklarheten sannsynligvis ble tvunget til eukaryoter, men at "de senere var i stand til å utnytte [det] til egen fordel." Bakterier og andre prokaryoter kan stole på veldefinert "digital" regulatorisk logikk fordi cellene deres har en tendens til å eksistere i bare noen få enkle, distinkte tilstander, for eksempel å bevege seg rundt og replikere.
Men de forskjellige celletilstandene for metazoer er "mye mer komplekse og noen ganger nær et kontinuum," sa Suter, så de er bedre tjent med uklare "analoge" reguleringer.
"Genreguleringssystemene i bakterier og eukaryoter ser ut til å ha divergert ganske betydelig," sa Tjian enig. Mens Monod sies å ha en gang bemerket at "hva er sant for E. coli er sant for elefanten,” ser det ut til at det ikke alltid er slik.
- SEO-drevet innhold og PR-distribusjon. Bli forsterket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk deg selv. Tilgang her.
- PlatoAiStream. Web3 Intelligence. Kunnskap forsterket. Tilgang her.
- PlatoESG. Karbon, CleanTech, Energi, Miljø, Solenergi, Avfallshåndtering. Tilgang her.
- PlatoHelse. Bioteknologisk og klinisk etterretning. Tilgang her.
- kilde: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- : har
- :er
- :ikke
- :hvor
- ][s
- $OPP
- 000
- 20
- 70
- a
- evne
- I stand
- Om oss
- fraværende
- rikelig
- akkumulert
- Handling
- skuespill
- Handling
- aktivere
- aktivitet
- handlinger
- tilpasse
- legge til
- la til
- ved siden av
- Fordel
- påvirke
- affinitet
- en gang til
- avtalte
- er enig
- algoritme
- Alle
- alene
- langs
- også
- Selv
- alltid
- blant
- forsterker
- an
- og
- dyr
- En annen
- noen
- hverandre
- ER
- argumentere
- oppstår
- rundt
- Artikkel
- AS
- aspektet
- antatt
- At
- oppmerksomhet
- forfatter
- Automatisert
- borte
- tilbake
- Bakterier
- basen
- BE
- fordi
- bli
- vært
- begynne
- være
- nytte
- Berkeley
- BEST
- Bedre
- mellom
- Stor
- Stort bilde
- større
- Biggest
- binde
- bindende
- biologi
- Blokker
- Blocks
- blåse
- øke
- både
- brakte
- men
- by
- california
- som heter
- kom
- CAN
- celle
- Celler
- viss
- utfordringer
- Endringer
- endring
- valg
- Kromosomer
- omstendigheter
- Classic
- Klassifisere
- fjerne
- klart
- Lukke
- tett
- Cluster
- samarbeide
- kollegaer
- samlet sett
- kombinasjon
- kombinasjoner
- Kom
- Utvalget
- Felles
- sammenligning
- komplekse
- komplisert
- komponent
- kompromiss
- forhold
- Tilkoblinger
- betraktninger
- innhold
- Kontinuum
- kontroll
- kontroller
- samtaler
- samarbeidende
- kunne
- Par
- mengde
- tiår
- bestemme
- avgjørelse
- avgjørelser
- Dekoding
- redusere
- avhengig
- avhenger
- dybde
- detalj
- detaljert
- Utvikling
- avvike
- forskjeller
- forskjellig
- vanskelig
- kringkasting
- digitalt
- digital mote
- direkte
- Funnet
- sykdom
- distinkt
- diverse
- dna
- do
- dock
- ikke
- domener
- Dominere
- ikke
- drevet
- dubbet
- under
- dynamisk
- Tidlig
- lett
- lett
- effekt
- Effektiv
- effekter
- enten
- elementer
- elefant
- andre steder
- dukke
- forbedre
- miljømessige
- feil
- Selv
- Hver
- bevis
- evolusjon
- utvikle seg
- utvikling
- nøyaktig
- eksempel
- eksempler
- eksisterer
- eksisterende
- forvente
- eksperimenter
- forklarte
- Exploit
- uttrykk
- ekstremt
- Faktisk
- faktor
- faktorer
- Mote
- Federal
- Noen få
- felt
- tenkte
- Finn
- Først
- passer
- fikset
- Til
- tvang
- skjema
- funnet
- Fransk
- fra
- funksjonelle
- funksjon
- funksjoner
- Gjeng
- samle
- samle
- sammenkomster
- generelt
- genomikk
- få
- blir
- gitt
- mål
- skal
- god
- styrende
- flott
- Gruppe
- gjette
- Halvparten
- håndtere
- skjedde
- Hard
- hardere
- Ha
- å ha
- he
- hjelper
- her
- svært
- hans
- hold
- Hvordan
- Men
- HTML
- HTTPS
- stort
- menneskelig
- Mennesker
- Tanken
- identifisert
- if
- Påvirkning
- in
- Inkludert
- faktisk
- individuelt
- påvirke
- å påvirke
- informasjon
- ubetydelig
- i stedet
- Institute
- samhandle
- samhandler
- interaksjoner
- inn
- innføre
- alltid
- involvere
- involvert
- innebærer
- IT
- DET ER
- selv
- jacob
- stikksag
- Jobb
- bare
- Hold
- holde
- Type
- slag
- Knowing
- lab
- stor
- Siste
- ledende
- læring
- Led
- mindre
- Life
- i likhet med
- Sannsynlig
- knyttet
- lite
- levende
- logikk
- Lang
- lenger
- så
- maskin
- maskinlæring
- laget
- magazine
- større
- gjøre
- GJØR AT
- måte
- mange
- massachusetts
- Massachusetts Institute of Technology
- Match
- materiale
- matematisk
- Saken
- Kan..
- kan være
- meningsfylt
- måle
- målinger
- mekanisme
- mekanismer
- medisinsk
- møter
- medlemmer
- metoder
- kunne
- MIT
- Mote
- Moduler
- molekyl~~POS=TRUNC
- molekyl
- mer
- flytte
- flytting
- mye
- flere
- må
- myriade
- mystisk
- Mystery
- Natur
- Nær
- nesten
- nødvendigvis
- Trenger
- nødvendig
- nettverk
- Nei.
- Merknader
- nå
- nyansert
- mange
- forekomme
- forekom
- forekommende
- of
- off
- tilby
- ofte
- on
- gang
- ONE
- bare
- or
- Annen
- vår
- ut
- Utfallet
- enn
- samlet
- egen
- pakket
- motstandere
- par
- Papir
- parametere
- del
- deltakere
- Spesielt
- Past
- Patches
- Betale
- kanskje
- overtalt
- fysisk
- bilde
- stykker
- Hvordan få de til å trives
- plato
- Platon Data Intelligence
- PlatonData
- Spille
- spillere
- Point
- posisjon
- mulig
- makt
- kraftig
- presis
- forutsi
- spådd
- tilstedeværelse
- presentere
- stolthet
- sannsynligvis
- prosedyren
- prosess
- produsere
- Produkt
- dyp
- programmer
- prosjekt
- Protein
- Proteiner
- gi
- gir
- beviser
- publisert
- sette
- puslespillet
- spørsmål
- raskt
- ganske
- tilfeldig
- Sats
- heller
- Raw
- lett
- klar
- ekte
- region
- regioner
- Regulere
- Regulering
- regulatorer
- relativt
- utgitt
- avhengige
- avhengig
- forbli
- bemerket
- fjerne
- reparasjon
- repeterende
- replikering
- forskere
- de
- Svare
- resultere
- resulterende
- Resultater
- Ris
- Rich
- ikke sant
- rigid
- Risiko
- Elvebredd
- RNA
- Rolle
- rom
- rutinemessig
- Kjør
- Sa
- samme
- sier
- forskere
- sikkert
- se
- synes
- tilsynelatende
- synes
- sett
- segmentet
- segmenter
- Gripe
- selektiv
- sensitive
- dømme
- September
- Sequence
- betjene
- servert
- flere
- Seksuell
- grunne
- Form
- Kort
- bør
- Vis
- betydning
- Enkelt
- siden
- enkelt
- nettstedet
- Nettsteder
- SIX
- Størrelse
- slakk
- mindre
- So
- løsning
- noen
- en eller annen måte
- noe
- noen ganger
- Kilder
- Rom
- spesifikk
- spesifisitet
- stabil
- stanford
- Stanford University
- Stater
- statistisk
- opphold
- Trinn
- stikker
- klebrig
- Stopp
- Story
- rett fram
- styrke
- sterk
- sterk
- strukturell
- struktur
- Student
- Studer
- I ettertid
- i det vesentlige
- slik
- sukker
- foreslår
- suite
- støtte
- sikker
- rundt
- Sveitsiske
- Bytte om
- sveits
- system
- Systemer
- Ta
- tandem
- Target
- mål
- Oppgave
- lag
- Teknologi
- forteller
- tendens
- titus
- enn
- Det
- De
- deres
- Dem
- seg
- deretter
- Der.
- derfor
- Disse
- de
- ting
- tror
- tenker
- denne
- De
- selv om?
- trodde
- tusener
- Gjennom
- hele
- Dermed
- tett
- tid
- ganger
- til
- sammen
- også
- Oversettelse
- gjennomsiktig
- fangst
- sant
- Turning
- to
- typen
- typer
- typisk
- ukr
- Til syvende og sist
- avdekket
- forstå
- universitet
- University of California
- ukjent
- upon
- bruke
- brukt
- nyttig
- vanligvis
- Verdier
- ulike
- enorme
- veldig
- levedyktig
- virus
- Voice
- venter
- var
- Se
- Vann
- Vei..
- måter
- we
- svak
- webp
- VI VIL
- veldefinerte
- Wells
- var
- Hva
- når
- om
- hvilken
- mens
- HVEM
- hele
- helt
- hvorfor
- vil
- med
- innenfor
- uten
- Arbeid
- arbeidet
- Verdens
- orm
- ville
- skrevet
- skrev
- ennå
- Du
- Young
- zephyrnet