Proteinele conduc multe procese biologice, cum ar fi activitatea enzimatică, transportul molecular și suportul celular. Structura tridimensională a unei proteine oferă o perspectivă asupra funcției sale și a modului în care interacționează cu alte biomolecule. Metodele experimentale pentru determinarea structurii proteinei, cum ar fi cristalografia cu raze X și spectroscopia RMN, sunt costisitoare și consumatoare de timp.
În schimb, metodele de calcul dezvoltate recent pot prezice rapid și precis structura unei proteine din secvența sa de aminoacizi. Aceste metode sunt critice pentru proteinele care sunt dificil de studiat experimental, cum ar fi proteinele membranare, ținta multor medicamente. Un exemplu binecunoscut în acest sens este AlphaFold, un algoritm bazat pe învățarea profundă, celebrat pentru predicțiile sale precise.
ESMFfold este o altă metodă foarte precisă, bazată pe învățarea profundă, dezvoltată pentru a prezice structura proteinelor din secvența sa de aminoacizi. ESMFold folosește un model de limbaj proteic mare (pLM) ca coloană vertebrală și funcționează cap la cap. Spre deosebire de AlphaFold2, nu are nevoie de o căutare sau Alinierea secvențelor multiple (MSA) și nici nu se bazează pe baze de date externe pentru a genera predicții. În schimb, echipa de dezvoltare a antrenat modelul pe milioane de secvențe de proteine de la UniRef. În timpul antrenamentului, modelul a dezvoltat modele de atenție care reprezintă în mod elegant interacțiunile evolutive dintre aminoacizii din secvență. Această utilizare a unui pLM în locul unui MSA permite timpi de predicție de până la 60 de ori mai rapid decât alte modele de ultimă generație.
În această postare, folosim modelul pre-antrenat ESMFold de la Hugging Face cu Amazon SageMaker pentru a prezice structura lanțului greu al trastuzumab, A anticorp monoclonal dezvoltat pentru prima dată de Genentech pentru tratamentul Cancer de sân HER2-pozitiv. Prezicerea rapidă a structurii acestei proteine ar putea fi utilă dacă cercetătorii ar dori să testeze efectul modificărilor secvenței. Acest lucru ar putea duce la o supraviețuire îmbunătățită a pacientului sau la mai puține efecte secundare.
Această postare oferă un exemplu de blocnotes Jupyter și scripturi aferente în cele ce urmează GitHub depozit.
Cerințe preliminare
Vă recomandăm să rulați acest exemplu într-un Amazon SageMaker Studio caiet rulează imaginea optimizată pentru CPU PyTorch 1.13 Python 3.9 pe un tip de instanță ml.r5.xlarge.
Vizualizați structura experimentală a trastuzumabului
Pentru început, folosim biopython bibliotecă și un script de ajutor pentru a descărca structura trastuzumab din Banca de date de proteine RCSB:
Apoi, folosim py3Dmol bibliotecă pentru a vizualiza structura ca o vizualizare 3D interactivă:
Următoarea figură reprezintă structura proteinei 3D 1N8Z din Protein Data Bank (PDB). În această imagine, lanțul ușor de trastuzumab este afișat în portocaliu, lanțul greu este albastru (cu regiunea variabilă în albastru deschis), iar antigenul HER2 este verde.
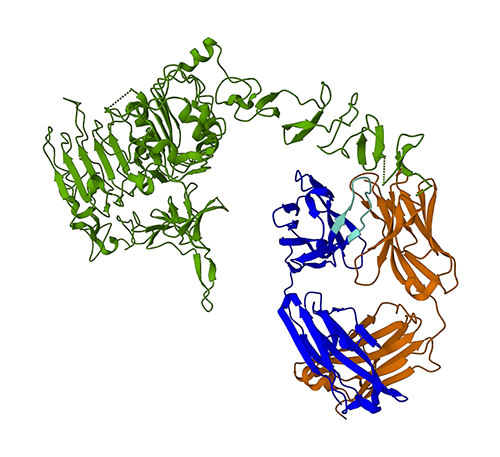
Vom folosi mai întâi ESMFold pentru a prezice structura lanțului greu (lanțul B) din secvența sa de aminoacizi. Apoi, vom compara predicția cu structura determinată experimental prezentată mai sus.
Preziceți structura lanțului greu de trastuzumab din secvența sa folosind ESMFold
Să folosim modelul ESMFold pentru a prezice structura lanțului greu și a o compara cu rezultatul experimental. Pentru început, vom folosi un mediu de notebook predefinit în Studio, care vine cu mai multe biblioteci importante, cum ar fi PyTorch, preinstalat. Deși am putea folosi un tip de instanță accelerată pentru a îmbunătăți performanța analizei notebook-ului nostru, vom folosi în schimb o instanță neaccelerată și vom rula predicția ESMFold pe un procesor.
În primul rând, încărcăm modelul ESMFold pre-antrenat și tokenizerul de la Hugging Face Hub:
Apoi, copiem modelul pe dispozitivul nostru (CPU în acest caz) și setăm câțiva parametri ai modelului:
Pentru a pregăti secvența de proteine pentru analiză, trebuie să o tokenizăm. Aceasta traduce simbolurile aminoacizilor (EVQLV…) într-un format numeric pe care modelul ESMFold îl poate înțelege (6,19,5,10,19,…):
Apoi, copiem intrarea tokenizată în mod, facem o predicție și salvăm rezultatul într-un fișier:
Acest lucru durează aproximativ 3 minute pe un tip de instanță neaccelerată, cum ar fi un r5.
Putem verifica acuratețea predicției ESMFold comparând-o cu structura experimentală. Facem acest lucru folosind US-Align instrument dezvoltat de Zhang Lab de la Universitatea din Michigan:
| PDBchain1 | PDBchain2 | TM-Scor |
| date/predicție.pdb:A | date/experimental.pdb:B | 0.802 |
scor de modelare șablon (TM-score) este o măsurătoare pentru evaluarea asemănării structurilor proteinelor. Un scor de 1.0 indică o potrivire perfectă. Scorurile de peste 0.7 indică faptul că proteinele au aceeași structură de coloană vertebrală. Scorurile de peste 0.9 indică faptul că proteinele sunt interschimbabile din punct de vedere funcțional pentru utilizare în aval. În cazul nostru de obținere a TM-Score 0.802, predicția ESMFold ar fi probabil adecvată pentru aplicații precum scorul structurii sau experimentele de legare a liganzilor, dar poate să nu fie potrivită pentru cazuri de utilizare precum înlocuire moleculară care necesită o precizie extrem de mare.
Putem valida acest rezultat prin vizualizarea structurilor aliniate. Cele două structuri prezintă un grad ridicat, dar nu perfect, de suprapunere. Predicțiile structurii proteinelor sunt un domeniu în evoluție rapidă și multe echipe de cercetare dezvoltă algoritmi din ce în ce mai precisi!
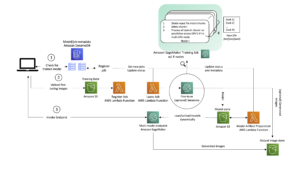
Implementați ESMFold ca punct final de inferență SageMaker
Rularea inferenței modelului într-un notebook este bine pentru experimentare, dar ce se întâmplă dacă trebuie să integrați modelul cu o aplicație? Sau o conductă MLOps? În acest caz, o opțiune mai bună este să vă implementați modelul ca punct final de inferență. În exemplul următor, vom implementa ESMFold ca punct final de inferență în timp real SageMaker pe o instanță accelerată. Punctele finale în timp real SageMaker oferă o modalitate scalabilă, rentabilă și sigură de a implementa și găzdui modele de învățare automată (ML). Cu scalarea automată, puteți ajusta numărul de instanțe care rulează punctul final pentru a satisface cerințele aplicației dvs., optimizând costurile și asigurând o disponibilitate ridicată.
Pre-construit Recipient SageMaker pentru Hugging Face facilitează implementarea modelelor de învățare profundă pentru sarcini comune. Cu toate acestea, pentru cazuri noi de utilizare, cum ar fi predicția structurii proteinelor, trebuie să definim un obicei inference.py script pentru a încărca modelul, a rula predicția și a formata rezultatul. Acest script include o mare parte din același cod pe care l-am folosit în notebook-ul nostru. De asemenea, creăm un requirements.txt fișier pentru a defini unele dependențe Python pe care punctul nostru final să le utilizeze. Puteți vedea fișierele pe care le-am creat în GitHub depozit.
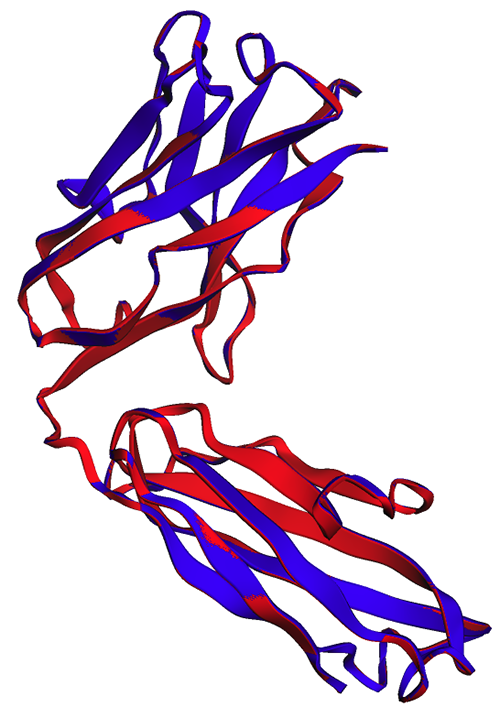
În figura următoare, structurile experimentale (albastru) și prezise (roșu) ale lanțului greu de trastuzumab sunt foarte asemănătoare, dar nu identice.

După ce am creat fișierele necesare în code director, implementăm modelul nostru folosind SageMaker HuggingFaceModel clasă. Aceasta folosește un container pre-construit pentru a simplifica procesul de implementare a modelelor Hugging Face în SageMaker. Rețineți că poate dura 10 minute sau mai mult pentru a crea punctul final, în funcție de disponibilitatea ml.g4dn tipuri de instanțe în Regiunea noastră.
Când implementarea punctului final este completă, putem retrimite secvența de proteine și afișa primele câteva rânduri ale predicției:
Deoarece am implementat punctul nostru final într-o instanță accelerată, predicția ar trebui să dureze doar câteva secunde. Fiecare rând din rezultat corespunde unui singur atom și include identitatea aminoacizilor, trei coordonate spațiale și o scorul pLDDT reprezentând încrederea de predicție în acea locație.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | OCUPARE | PLDDT | ATOM_ID |
| ATOM | 1 | N | G.L.U. | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | G.L.U. | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | G.L.U. | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | G.L.U. | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | G.L.U. | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | G.L.U. | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | G.L.U. | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | G.L.U. | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | G.L.U. | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Folosind aceeași metodă ca înainte, vedem că predicțiile pentru notebook și punctul final sunt identice.
| PDBchain1 | PDBchain2 | TM-Scor |
| data/endpoint_prediction.pdb:A | date/predicție.pdb:A | 1.0 |
După cum se observă în figura următoare, predicțiile ESMFold generate în notebook (roșu) și de la punctul final (albastru) arată o aliniere perfectă.
A curăța
Pentru a evita alte taxe, ștergem punctul final de inferență și datele de testare:
Rezumat
Predicția computațională a structurii proteinelor este un instrument critic pentru înțelegerea funcției proteinelor. Pe lângă cercetarea de bază, algoritmi precum AlphaFold și ESMFold au multe aplicații în medicină și biotehnologie. Perspectivele structurale generate de aceste modele ne ajută să înțelegem mai bine modul în care biomoleculele interacționează. Acest lucru poate duce apoi la instrumente de diagnostic și terapii mai bune pentru pacienți.
În această postare, arătăm cum să implementăm modelul de limbaj de proteine ESMFold de la Hugging Face Hub ca punct final de inferență scalabil folosind SageMaker. Pentru mai multe informații despre implementarea modelelor Hugging Face pe SageMaker, consultați Utilizați Hugging Face cu Amazon SageMaker. De asemenea, puteți găsi mai multe exemple de știință a proteinelor în Analiză uimitoare a proteinelor pe AWS Repoziție GitHub. Vă rugăm să ne lăsați un comentariu dacă există alte exemple pe care ați dori să le vedeți!
Despre Autori
 Brian Loyal este arhitect senior de soluții AI/ML în echipa Global Healthcare și Life Sciences de la Amazon Web Services. Are o experiență de peste 17 ani în biotehnologie și învățare automată și este pasionat de a ajuta clienții să rezolve provocările genomice și proteomice. În timpul liber, îi place să gătească și să mănânce cu prietenii și familia.
Brian Loyal este arhitect senior de soluții AI/ML în echipa Global Healthcare și Life Sciences de la Amazon Web Services. Are o experiență de peste 17 ani în biotehnologie și învățare automată și este pasionat de a ajuta clienții să rezolve provocările genomice și proteomice. În timpul liber, îi place să gătească și să mănânce cu prietenii și familia.
 Shamika Ariyawansa este arhitect specializat în soluții AI/ML în echipa Global Healthcare și Life Sciences de la Amazon Web Services. Lucrează cu pasiune cu clienții pentru a-și accelera adoptarea AI și ML, oferind îndrumări tehnice și ajutându-i să inoveze și să construiască soluții cloud sigure pe AWS. În afara serviciului, iubește schiul și off-roading-ul.
Shamika Ariyawansa este arhitect specializat în soluții AI/ML în echipa Global Healthcare și Life Sciences de la Amazon Web Services. Lucrează cu pasiune cu clienții pentru a-și accelera adoptarea AI și ML, oferind îndrumări tehnice și ajutându-i să inoveze și să construiască soluții cloud sigure pe AWS. În afara serviciului, iubește schiul și off-roading-ul.
 Yanjun Qi este manager senior de știință aplicată la Laboratorul de soluții de învățare automată AWS. Ea inovează și aplică învățarea automată pentru a ajuta clienții AWS să-și accelereze adoptarea AI și a cloud-ului.
Yanjun Qi este manager senior de știință aplicată la Laboratorul de soluții de învățare automată AWS. Ea inovează și aplică învățarea automată pentru a ajuta clienții AWS să-și accelereze adoptarea AI și a cloud-ului.
- Distribuție de conținut bazat pe SEO și PR. Amplifică-te astăzi.
- PlatoAiStream. Web3 Data Intelligence. Cunoștințe amplificate. Accesați Aici.
- Mintând viitorul cu Adryenn Ashley. Accesați Aici.
- Cumpărați și vindeți acțiuni în companii PRE-IPO cu PREIPO®. Accesați Aici.
- Sursa: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :are
- :este
- :nu
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Despre Noi
- mai sus
- AC
- accelera
- accelerat
- precizie
- precis
- precis
- realizarea
- activitate
- plus
- Adoptare
- AI
- AI / ML
- Algoritmul
- algoritmi
- aliniat
- aliniere
- de asemenea
- Cu toate ca
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- analiză
- și
- O alta
- Orice
- aplicație
- aplicatii
- aplicat
- adecvat
- SUNT
- AS
- evaluarea
- At
- atom
- atenţie
- Automat
- disponibilitate
- evita
- AWS
- Învățare automată AWS
- Șira spinării
- Bancă
- de bază
- BE
- înainte
- începe
- Mai bine
- între
- legare
- biotehnologie
- Albastru
- construi
- dar
- by
- CAN
- Rac
- desen animat
- caz
- cazuri
- celebru
- lanţ
- provocări
- taxe
- verifica
- clasă
- Cloud
- adoptarea norului
- cod
- culoare
- vine
- comentariu
- Comun
- comparaţie
- compararea
- Completă
- încredere
- Recipient
- contrast
- corespunde
- cost-eficiente
- Cheltuieli
- ar putea
- crea
- a creat
- critic
- clienţii care
- de date
- baze de date
- datetime
- adânc
- învățare profundă
- DeepMind
- Grad
- cererile
- În funcție
- implementa
- dislocate
- Implementarea
- desfășurarea
- Determina
- determinat
- dezvoltat
- în curs de dezvoltare
- Dezvoltare
- dispozitiv
- dificil
- Afişa
- do
- face
- Nu
- Descarca
- conduce
- Droguri
- în timpul
- fiecare
- uşor
- efect
- efecte
- permite
- capăt
- Punct final
- asigurare
- Mediu inconjurator
- exemplu
- exemple
- scump
- experienţă
- experimente
- extern
- extrem
- Față
- familie
- mai repede
- puțini
- mai puține
- camp
- Figura
- Fișier
- Fişiere
- Găsi
- capăt
- First
- următor
- Pentru
- format
- Prietenii lui
- din
- funcţie
- mai mult
- genera
- generată
- GitHub
- Caritate
- Verde
- îndrumare
- Avea
- he
- de asistență medicală
- greu
- ajutor
- ajutor
- Înalt
- lui
- gazdă
- Cum
- Cum Pentru a
- Totuși
- HTML
- HTTPS
- Butuc
- Față îmbrățișată
- identic
- Identitate
- if
- imagine
- import
- important
- îmbunătăţi
- îmbunătățit
- in
- include
- indica
- indică
- informații
- inova
- intrare
- înţelegere
- perspective
- instanță
- in schimb
- integra
- interacţiona
- interacţiuni
- interactiv
- interactiv
- în
- IT
- ESTE
- de laborator
- limbă
- mare
- conduce
- învăţare
- Părăsi
- biblioteci
- Bibliotecă
- Viaţă
- Life Sciences
- ușoară
- ca
- Probabil
- încărca
- locaţie
- căutare
- maşină
- masina de învățare
- face
- FACE
- manager
- multe
- Meci
- Mai..
- medicină
- Întâlni
- metodă
- Metode
- metric
- Michigan
- milioane
- minute
- ML
- MLOps
- mod
- model
- Modele
- modificările aduse
- molecular
- mai mult
- mult
- nume
- necesar
- Nevoie
- NIH
- caiet
- roman
- număr
- obiecte
- învechit
- of
- on
- ONE
- afară
- opereaza
- optimizarea
- Opțiune
- or
- Portocaliu
- OS
- Altele
- al nostru
- producție
- exterior
- parametrii
- pasionat
- pacient
- pacientes
- modele
- Perfect
- performanță
- conducte
- Plato
- Informații despre date Platon
- PlatoData
- "vă rog"
- Post
- potenţial
- prezice
- a prezis
- estimarea
- prezicere
- Predictii
- Pregăti
- în prealabil
- proces
- procese
- Proteină
- Proteine
- furniza
- furnizează
- furnizarea
- Piton
- pirtorh
- Qi
- repede
- repede
- domeniu în evoluție rapidă
- în timp real
- recomanda
- Roșu
- regiune
- legate de
- se bazează
- reprezenta
- reprezentând
- reprezintă
- necesita
- cercetare
- cercetători
- rezultat
- RÂND
- Alerga
- funcţionare
- sagemaker
- SageMaker Inference
- acelaşi
- Economisiți
- scalabil
- scalare
- Ştiinţă
- ȘTIINȚE
- scor
- notare
- script-uri
- secunde
- sigur
- vedea
- senior
- Secvenţă
- Servicii
- set
- câteva
- Distribuie
- ea
- să
- Arăta
- indicat
- parte
- asemănător
- simplifica
- singur
- soluţie
- soluţii
- REZOLVAREA
- unele
- spațial
- specialist
- Spectroscopie
- viteză
- Începe
- de ultimă oră
- Pas
- structural
- structura
- studio
- Studiu
- astfel de
- potrivit
- a sustine
- supravieţuire
- Lua
- ia
- obiective
- sarcini
- echipă
- echipe
- Tehnic
- test
- decât
- acea
- lor
- Lor
- apoi
- Acolo.
- Acestea
- acest
- trei
- tri-dimensională
- timp
- consumă timp
- ori
- la
- tokeniza
- cuvinte pot
- instrument
- Unelte
- lanternă
- dresat
- Pregătire
- transformatoare
- de transport
- tratament
- Două
- tip
- Tipuri
- înţelege
- înţelegere
- universitate
- spre deosebire de
- us
- utilizare
- utilizat
- folosind
- VALIDA
- foarte
- Vizualizare
- vizualizare
- W
- dorit
- Cale..
- we
- web
- servicii web
- bine cunoscut
- Ce
- Wikipedia
- voi
- cu
- WordPress
- Apartamente
- fabrică
- ar
- radiografie
- Tu
- Ta
- zephyrnet